안녕하세요! Multi-omics 데이터 분석에 이제 막 관심을 갖기 시작한 분들을 위해, LLM(Large Language Model)을 보조 도구로 활용하여 Multi-omics 분석의 기초를 체험해보는 최소한의 과정을 안내해 드립니다. 이 가이드는 복잡한 통계나 전문 분석 도구 설치 없이, LLM과의 대화를 통해 개념을 이해하고 간단한 데이터 탐색을 경험하는 데 초점을 맞춥니다.
🎯 목표:
- Multi-omics 데이터가 무엇인지 감 잡기
- LLM을 활용하여 데이터 탐색 아이디어 얻기
- LLM으로 간단한 분석 코드 생성 및 실행 맛보기
- LLM을 통해 분석 결과(가상) 해석 도움받기
✨ 준비물:
- 웹 브라우저: 인터넷 접속 가능한 환경
- LLM 서비스 접속 계정: ChatGPT, Google Gemini, Claude 등 선호하는 LLM 서비스
- (선택) Google Colab 또는 로컬 Python 환경: 간단한 코드 실행을 위해 (없어도 LLM과의 대화만으로 진행 가능)
🚀 1단계: Multi-omics 세상 살짝 엿보기 (LLM과 대화하기)
Multi-omics 분석은 여러 종류의 생물학적 데이터(유전체, 전사체, 단백체 등)를 통합하여 생명 현상을 더 깊이 이해하려는 접근 방식입니다. 먼저 LLM에게 Multi-omics가 무엇인지, 왜 중요한지 물어보며 감을 잡아봅시다.
💬 LLM에게 질문해보세요:
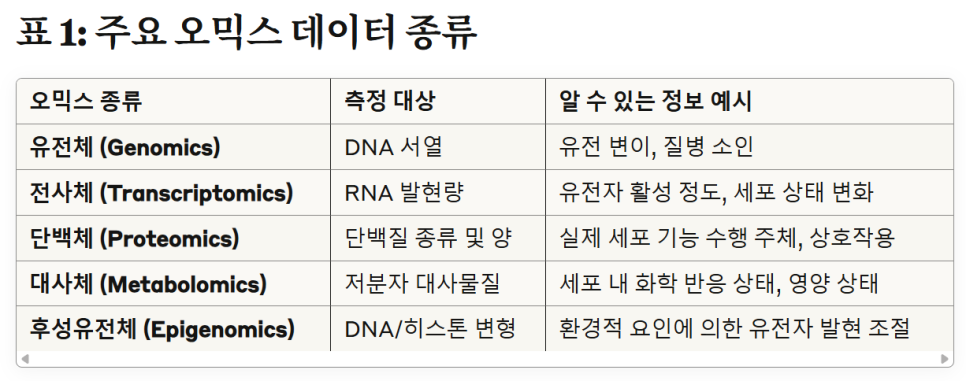
1. 멀티오믹스(Multi-omics) 분석이 뭐야? 초등학생도 이해할 수 있게 쉽게 설명해줘. 2. 멀티오믹스 분석은 왜 중요해? 어떤 장점이 있어? 3. 멀티오믹스 분석에 주로 사용되는 데이터 종류는 뭐가 있어? 표로 정리해줄래?
🤖 LLM 예상 답변 (예시):
멀티오믹스 분석이란? 우리 몸이나 세포 같은 생명체를 아주 자세히 들여다보는 방법이에요. 마치 한 사람을 알기 위해 그 사람의 일기장(유전체), 주고받은 편지(전사체), 평소 먹는 음식(대사체), 건강 검진 기록(단백체) 등을 모두 모아서 종합적으로 살펴보는 것과 같아요. 각 정보(오믹스 데이터)만 따로 볼 때보다 훨씬 더 정확하고 깊게 그 사람(생명 현상)을 이해할 수 있게 되죠.
중요성과 장점:
더 깊은 이해: 생명 현상의 복잡한 원리를 다각도에서 파악할 수 있어요.
새로운 발견: 질병의 원인을 찾거나 새로운 치료법을 개발하는 데 중요한 단서를 제공할 수 있어요.
맞춤 의학: 개인별 데이터 특성에 맞는 정밀한 진단과 치료 전략을 세우는 데 도움을 줄 수 있어요.
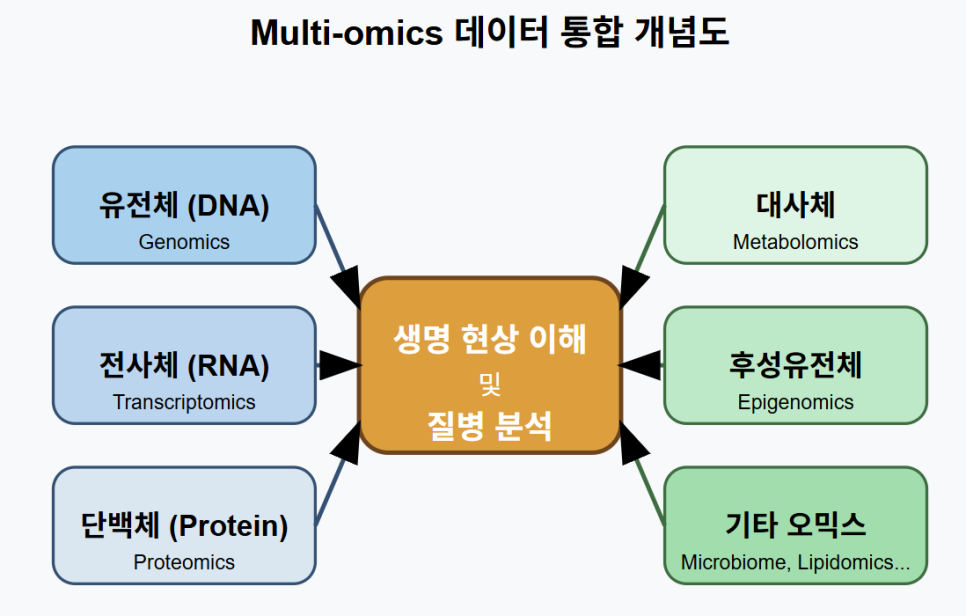
[그림 1: Multi-omics 데이터 통합 개념도]
🚀 2단계: 가상의 Multi-omics 데이터 상상하고 LLM과 탐색 계획 세우기
실제 Multi-omics 데이터는 매우 크고 복잡합니다. 여기서는 아주 간단한 가상의 데이터를 상상하고, 이 데이터를 어떻게 탐색해볼 수 있을지 LLM과 함께 아이디어를 구체화해 보겠습니다.
상황 설정: 암 환자 5명에게서 종양 조직을 얻어, **유전자 발현 데이터(RNA)**와 **단백질 발현 데이터(Protein)**를 측정했다고 가정해 봅시다.
- 데이터 형태 (상상):
- RNA_data.csv: 환자별(행) 특정 유전자들(열)의 발현량 값
- Protein_data.csv: 환자별(행) 특정 단백질들(열)의 발현량 값
💬 LLM에게 질문해보세요:
암 환자 5명의 RNA 데이터와 Protein 데이터가 있다고 가정해볼게. 각 데이터는 환자별 유전자/단백질 발현량을 담고 있어. 1. 이 두 종류의 데이터를 가지고 어떤 분석을 해볼 수 있을까? 분석 아이디어 목록을 만들어줘. 2. 각 분석 아이디어에 대해 어떤 정보를 얻을 수 있는지 간단히 설명해줘. 3. 만약 특정 유전자 A의 발현량과 특정 단백질 B의 발현량 사이에 양의 상관관계가 나타난다면, 이걸 어떻게 해석해 볼 수 있을까? 가능한 생물학적 의미를 몇 가지 알려줘.
🤖 LLM 예상 답변 (예시):
분석 아이디어 목록:
개별 오믹스 데이터 탐색:
RNA 데이터 내에서 환자별로 발현량이 높은 유전자 찾기
Protein 데이터 내에서 환자별로 발현량이 높은 단백질 찾기
환자 간 유전자/단백질 발현 패턴 비교 (예: 특정 그룹에서만 높게 발현되는 유전자/단백질)
Multi-omics 데이터 통합 탐색:
상관관계 분석: 특정 유전자 발현량과 특정 단백질 발현량 간의 상관관계 계산 (예: 유전자 A와 단백질 B의 발현량이 함께 증가/감소하는가?)
패턴 매칭: RNA 데이터에서의 환자 그룹 패턴과 Protein 데이터에서의 환자 그룹 패턴 비교
(고급) 통합 군집 분석: RNA와 Protein 데이터를 함께 사용하여 환자들을 유사한 그룹으로 묶기
아이디어별 얻을 수 있는 정보:
개별 탐색: 각 데이터 종류 내에서의 주요 특징이나 환자 간 차이를 파악할 수 있습니다. 특정 암 아형(subtype)과 관련된 유전자/단백질 후보를 찾을 수 있습니다.
통합 탐색: 유전자 발현 조절이 실제 단백질 수준 변화로 이어지는지(상관관계 분석), 서로 다른 데이터 타입에서 일관된 생물학적 패턴이 나타나는지(패턴 매칭, 군집 분석) 등을 파악하여 더 신뢰도 높은 해석을 할 수 있습니다.
유전자 A - 단백질 B 양의 상관관계 해석:
직접 조절: 유전자 A가 단백질 B를 직접 암호화하고 있을 가능성 (가장 흔함). 유전자 발현이 증가하면 해당 단백질 합성도 증가.
간접 조절: 유전자 A가 단백질 B의 발현을 조절하는 다른 인자(예: 전사 인자)를 활성화시킬 가능성.
공통 조절: 유전자 A와 단백질 B가 동일한 상위 조절 인자나 신호 전달 경로에 의해 함께 조절될 가능성.
기능적 연관성: 유전자 A와 단백질 B가 동일한 생물학적 과정이나 복합체 내에서 함께 기능할 가능성.
[표 2: 가상 데이터 구조 예시]
RNA_data.csv (예시)
|
Patient
|
Gene_X
|
Gene_Y
|
Gene_Z
|
|
P1
|
10.5
|
2.1
|
8.8
|
|
P2
|
12.1
|
1.5
|
9.5
|
|
P3
|
3.2
|
5.8
|
4.1
|
|
P4
|
11.8
|
1.9
|
9.2
|
|
P5
|
4.5
|
6.2
|
3.8
|
Protein_data.csv (예시)
|
Patient
|
Protein_A
|
Protein_B
|
Protein_C
|
|
P1
|
50.2
|
105.1
|
30.5
|
|
P2
|
55.8
|
110.8
|
28.1
|
|
P3
|
20.1
|
45.2
|
80.9
|
|
P4
|
53.5
|
108.3
|
31.2
|
|
P5
|
25.3
|
50.1
|
75.4
|
🚀 3단계: LLM 도움 받아 간단한 코드 실행해보기 (Python & Pandas)
이제 LLM에게 간단한 데이터 분석 코드를 생성해달라고 요청하고, (가능하다면) Google Colab 등에서 직접 실행해 봅시다. 여기서는 위 가상 데이터를 Python의 pandas 라이브러리를 이용해 만들고, 기본적인 탐색을 수행하는 코드를 요청합니다.
💬 LLM에게 요청해보세요:
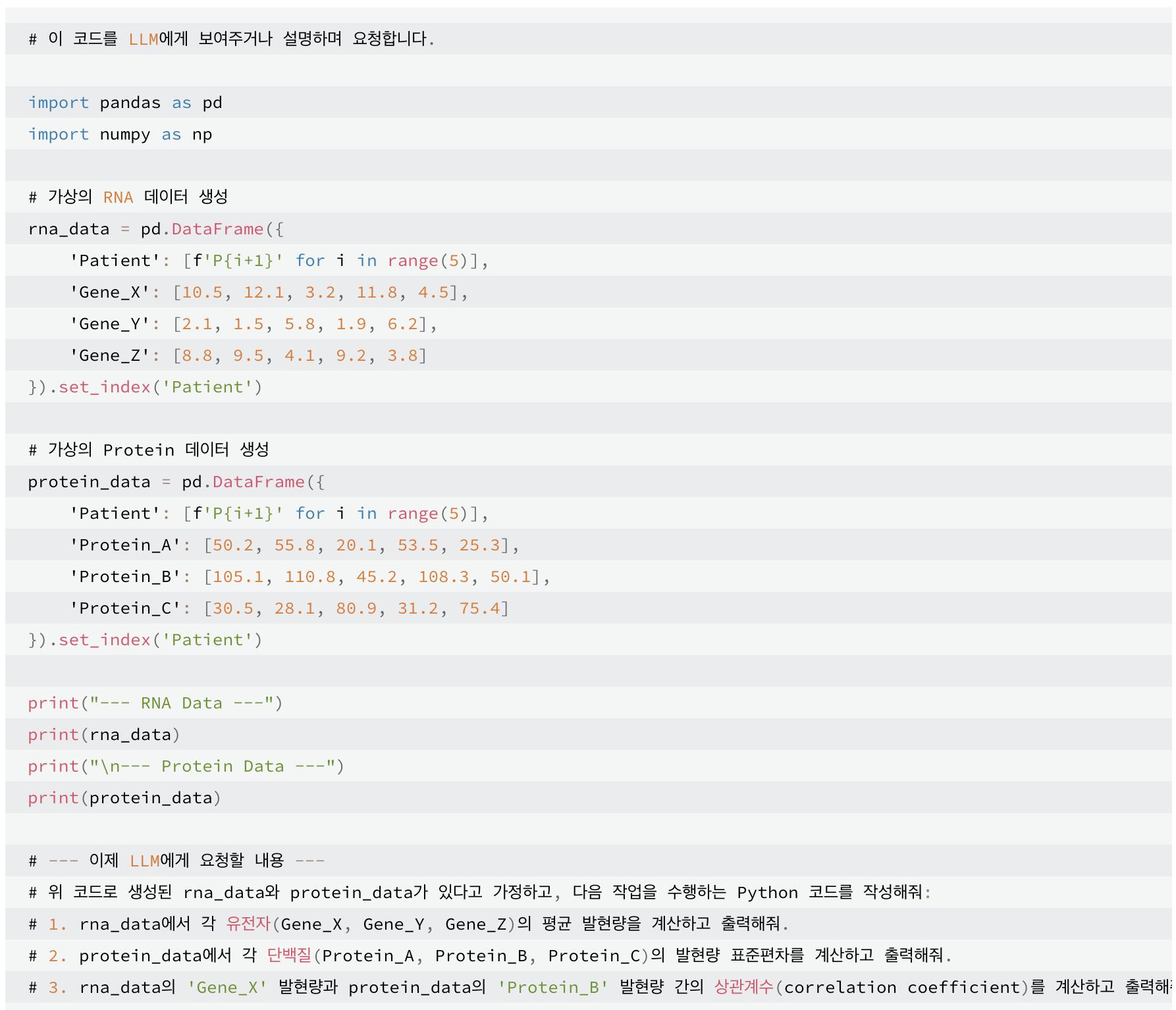
# 이 코드를 LLM에게 보여주거나 설명하며 요청합니다.
import pandas as pd
import numpy as np
# 가상의 RNA 데이터 생성
rna_data = pd.DataFrame({
'Patient': [f'P{i+1}' for i in range(5)],
'Gene_X': [10.5, 12.1, 3.2, 11.8, 4.5],
'Gene_Y': [2.1, 1.5, 5.8, 1.9, 6.2],
'Gene_Z': [8.8, 9.5, 4.1, 9.2, 3.8]
}).set_index('Patient')
# 가상의 Protein 데이터 생성
protein_data = pd.DataFrame({
'Patient': [f'P{i+1}' for i in range(5)],
'Protein_A': [50.2, 55.8, 20.1, 53.5, 25.3],
'Protein_B': [105.1, 110.8, 45.2, 108.3, 50.1],
'Protein_C': [30.5, 28.1, 80.9, 31.2, 75.4]
}).set_index('Patient')
print("--- RNA Data ---")
print(rna_data)
print("\n--- Protein Data ---")
print(protein_data)
# --- 이제 LLM에게 요청할 내용 ---
# 위 코드로 생성된 rna_data와 protein_data가 있다고 가정하고, 다음 작업을 수행하는 Python 코드를 작성해줘:
# 1. rna_data에서 각 유전자(Gene_X, Gene_Y, Gene_Z)의 평균 발현량을 계산하고 출력해줘.
# 2. protein_data에서 각 단백질(Protein_A, Protein_B, Protein_C)의 발현량 표준편차를 계산하고 출력해줘.
# 3. rna_data의 'Gene_X' 발현량과 protein_data의 'Protein_B' 발현량 간의 상관계수(correlation coefficient)를 계산하고 출력해줘.
🤖 LLM 예상 생성 코드 (예시):
# LLM이 생성해 줄 것으로 기대되는 코드
# 1. RNA 데이터 유전자별 평균 발현량 계산
rna_means = rna_data.mean(axis=0) # axis=0 은 열(유전자) 기준 계산
print("\n--- RNA Mean Expression ---")
print(rna_means)
# 2. Protein 데이터 단백질별 발현량 표준편차 계산
protein_stds = protein_data.std(axis=0) # axis=0 은 열(단백질) 기준 계산
print("\n--- Protein Expression Standard Deviation ---")
print(protein_stds)
# 3. Gene_X와 Protein_B 발현량 간 상관계수 계산
# 먼저 두 시리즈(열)를 추출합니다.
gene_x_expression = rna_data['Gene_X']
protein_b_expression = protein_data['Protein_B']
# 상관계수를 계산합니다.
correlation = gene_x_expression.corr(protein_b_expression)
print("\n--- Correlation between Gene_X and Protein_B ---")
print(f"Correlation coefficient: {correlation:.4f}")
# (추가) 간단한 시각화 코드 요청도 가능합니다 (matplotlib/seaborn 사용)
# 예: "Gene_X와 Protein_B의 발현량을 점으로 찍는 scatter plot을 그려주는 코드도 만들어줘."
import matplotlib.pyplot as plt
import seaborn as sns
plt.figure(figsize=(6, 4)) sns.scatterplot(x=gene_x_expression, y=protein_b_expression)
plt.title('Gene_X vs Protein_B Expression')
plt.xlabel('Gene_X Expression')
plt.ylabel('Protein_B Expression')
# 각 점에 환자 이름 표시 (옵션)
for i, patient in enumerate(rna_data.index):
plt.text(gene_x_expression[i], protein_b_expression[i], patient)
plt.grid(True)
plt.show()
💻 실행: 위 Python 코드를 Google Colab의 코드 셀이나 로컬 Python 환경에 붙여넣고 실행해 보세요. pandas 라이브러리가 없다면 pip install pandas (Colab에서는 !pip install pandas) 명령으로 설치해야 합니다. 시각화 코드를 실행하려면 matplotlib과 seaborn도 필요할 수 있습니다 (pip install matplotlib seaborn).
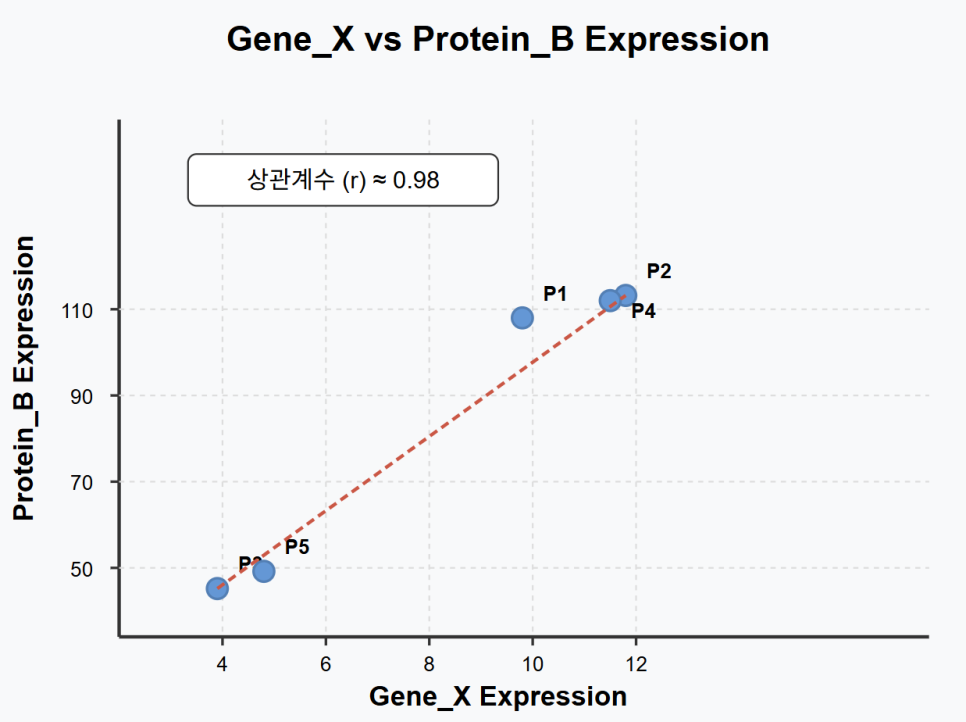
[그림 2: 생성된 Scatter Plot 예시]
🚀 4단계: LLM과 함께 결과 해석하고 다음 단계 구상하기
코드 실행 결과(평균, 표준편차, 상관계수, 그래프 등)를 바탕으로 LLM에게 다시 질문하며 의미를 파악하고, 더 나아가 어떤 분석을 해볼 수 있을지 아이디어를 얻어봅니다.
💬 LLM에게 질문해보세요:
# 이전 단계에서 얻은 결과(예: 상관계수 값, 그래프 모양)를 설명하며 질문합니다. 1. Gene_X와 Protein_B의 상관계수가 약 0.98로 매우 높게 나왔어. 아까 네가 설명해준 해석 가능성 중에서 어떤 것이 가장 유력해 보일까? 그 이유는 뭐야? 2. RNA 데이터에서 Gene_Y는 평균 발현량이 낮은데, Protein 데이터에서 Protein_C는 변동성(표준편차)이 꽤 크게 나왔어. 이런 경우 어떤 추가적인 분석이나 확인을 해보면 좋을까? 아이디어를 몇 가지 제시해줘. 3. 이 가상 데이터 분석 경험을 바탕으로, 실제 Multi-omics 연구를 시작하려면 어떤 것들을 더 배우거나 준비해야 할까? 학습 로드맵이나 필요한 기술 스택을 알려줘.
🤖 LLM 예상 답변 (예시):
Gene_X와 Protein_B의 높은 상관관계 해석:
상관계수가 0.98로 매우 높다면, **"유전자 A가 단백질 B를 직접 암호화"**하는 경우가 가장 유력해 보입니다. 유전자 발현(전사) 수준과 단백질 발현 수준이 이렇게까지 강하게 비례하는 것은 보통 직접적인 연결 고리가 있을 때 나타나는 현상입니다. 물론 이것은 가상 데이터에서의 결과이며, 실제 데이터에서는 실험적 검증이 필요합니다.
Gene_Y(낮은 평균)와 Protein_C(높은 변동성) 관련 추가 분석 아이디어:
전사 후 조절(Post-transcriptional regulation) 확인: Gene_Y RNA가 안정적이지 않거나, 번역 효율이 환자마다 크게 다를 수 있습니다. 관련 microRNA나 RNA 결합 단백질 데이터를 추가로 확인해 볼 수 있습니다.
단백질 분해(Protein degradation) 속도 차이 확인: Protein_C의 안정성이 환자마다 달라, 생성량은 비슷해도 분해 속도 차이로 인해 최종 양의 변동성이 커질 수 있습니다. 단백질 분해 관련 경로(예: 유비퀴틴-프로테아좀 시스템) 관련 데이터 분석을 고려해 볼 수 있습니다.
측정 오류 가능성 검토: RNA 측정값이나 Protein 측정값 자체에 오류나 배치 효과(batch effect)가 있었을 가능성도 배제할 수 없습니다. 데이터 품질 관리(QC) 과정을 다시 확인해야 합니다.
다른 오믹스 데이터와의 연관성 분석: 혹시 특정 유전형(Genomics)이나 후성유전적 변이(Epigenomics)가 Protein_C의 발현 변동성과 관련 있는지 확인해 볼 수 있습니다.
실제 Multi-omics 연구를 위한 학습 로드맵/기술 스택:
기초 다지기:
생물학: 분자생물학, 유전학, 세포생물학 기본 지식 (각 오믹스 데이터의 의미 이해)
통계학: 기초 통계, 가설 검정, 다변량 분석, 기계 학습 기본 원리
프로그래밍: Python (Pandas, Numpy, Scipy, Matplotlib, Seaborn 필수) 또는 R (tidyverse, Bioconductor 패키지 활용) 능력
핵심 기술 습득:
각 오믹스 데이터 분석: NGS 데이터 처리 파이프라인(RNA-seq, WGS/WES 등), 단백체 데이터 분석(MaxQuant 등) 경험
Multi-omics 통합 분석 도구: MOFA+, Seurat (WNN), MultiAssayExperiment 등 전문 라이브러리/패키지 사용법 학습
데이터 시각화: 복잡한 데이터를 효과적으로 표현하는 방법 (heatmap, network visualization, dimensionality reduction plots 등)
경로 분석/기능 주석: 유전자/단백질 목록의 생물학적 의미 해석 (GO enrichment, KEGG pathway analysis 등)
심화 학습:
고급 통계/기계 학습: 차원 축소(PCA, UMAP), 군집 분석(Clustering), 분류(Classification), 네트워크 분석, 인과관계 추론 등
클라우드 컴퓨팅/빅데이터 처리: 대용량 데이터 분석을 위한 환경 (AWS, GCP, Azure) 경험
최신 연구 동향 팔로우: 관련 논문, 컨퍼런스 참여

🚀 다음 단계
- 관심 있는 특정 오믹스 데이터(예: RNA-seq) 분석 튜토리얼 따라 해보기
- Python 또는 R 프로그래밍 및 관련 생물정보학 라이브러리 학습 시작하기
- MOFA+, Seurat 등 실제 Multi-omics 통합 분석 툴 사용법 익히기
- The Cancer Genome Atlas (TCGA) 등 공개된 실제 Multi-omics 데이터셋 탐색해보기 (데이터 접근 및 분석 난이도 높음)
이 가이드가 Multi-omics 분석의 흥미로운 세계로 나아가는 첫걸음이 되기를 응원합니다!